前言
2020 年全球的关键词非COVID19 莫属。虽然现在关于病毒的起源众说纷纭,也引起了不小的外交冲突。作为数据爱好者,还是用数据说话比较靠谱。
COVID19数据来源有很多,这里仅仅选kaggle上的数据,链接如下:https://www.kaggle.com/sudalairajkumar/novel-corona-virus-2019-dataset。 这里面的数据会持续更新,所以你拿到的数据可能会和我不同。
该链接共包含以下数据集:
- COVID19_line_list_data.csv(358.85 KB)--> 关于一些每次确诊个例的报告
- COVID19_open_line_list.csv(2.93 MB)--> 更详细的确诊个例报告
- covid_19_data.csv(1.53 MB)--> 各国确诊数据,时间线为行
- time_series_covid_19_confirmed.csv(100.3 KB)--> 时间线为列的各国确诊数据
- time_series_covid_19_confirmed_US.csv(1.11 MB)--> 美国确诊相关的数据
- time_series_covid_19_deaths_US.csv(1.04 MB)--> 美国死亡数据
- time_series_covid_19_deaths.csv(76.09 KB)--> 时间线为列的各国死亡数据
- time_series_covid_19_recovered.csv(84.62 KB)-->时间线为列的治愈人数数据
各个数据集的侧重点不同,今天我们分析一下第一组数据,COVID19_line_list_data。

加载数据
首先还是加载一些包,我首先预计会用到这几个包,后面用的包会在后面导入。
import plotly.graph_objects as go
from collections import Counter
import missingno as msno
import pandas as pd数据源我已经提前下好,并且放到代码所在路径的data 文件中,你可以根据你的情况调整路径。
line_list_data_file = 'data/COVID19_line_list_data.csv'一如既往,首先查看数据统计信息。
line_list_data_raw_df = pd.read_csv(line_list_data_file)
print(line_list_data_raw_df.info())
print(line_list_data_raw_df.describe())结果如下,系统识别出了27列的数据,但是仔细看,有多列数据Non-Null Count 为0,意味着为空列,样本量为1085行。
Backend TkAgg is interactive backend. Turning interactive mode on.
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 1085 entries, 0 to 1084
Data columns (total 27 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 id 1085 non-null int64
1 case_in_country 888 non-null float64
2 reporting date 1084 non-null object
3 Unnamed: 3 0 non-null float64
4 summary 1080 non-null object
5 location 1085 non-null object
6 country 1085 non-null object
7 gender 902 non-null object
8 age 843 non-null float64
9 symptom_onset 563 non-null object
10 If_onset_approximated 560 non-null float64
11 hosp_visit_date 507 non-null object
12 exposure_start 128 non-null object
13 exposure_end 341 non-null object
14 visiting Wuhan 1085 non-null int64
15 from Wuhan 1081 non-null float64
16 death 1085 non-null object
17 recovered 1085 non-null object
18 symptom 270 non-null object
19 source 1085 non-null object
20 link 1085 non-null object
21 Unnamed: 21 0 non-null float64
22 Unnamed: 22 0 non-null float64
23 Unnamed: 23 0 non-null float64
24 Unnamed: 24 0 non-null float64
25 Unnamed: 25 0 non-null float64
26 Unnamed: 26 0 non-null float64
dtypes: float64(11), int64(2), object(14)
memory usage: 229.0+ KB
None
id case_in_country ... Unnamed: 25 Unnamed: 26
count 1085.000000 888.000000 ... 0.0 0.0
mean 543.000000 48.841216 ... NaN NaN
std 313.356825 78.853528 ... NaN NaN
min 1.000000 1.000000 ... NaN NaN
25% 272.000000 11.000000 ... NaN NaN
50% 543.000000 28.000000 ... NaN NaN
75% 814.000000 67.250000 ... NaN NaN
max 1085.000000 1443.000000 ... NaN NaN
[8 rows x 13 columns]删除空列
pandas 提供了方便的dropna 函数,可以识别出所有的nan 数据,并且标识为True,Dataframe 可以对每列(axis=1)的所有布尔标识进行逻辑运算(any 或者是all),相当于or 或者and 运算,之后得到1维的标识,进行删除。 个人习惯于对一个dataframe 直接操作,可以节省变量内存,因此后续很多操作都会设置inplace=True。
line_list_data_raw_df.dropna(axis=1, how='all', inplace=True)
print(f'df shape is {line_list_data_raw_df.shape}')
df shape is (1085, 20)数据缺失可视化
缺失值查询很简单,用info函数很容易得到统计数据,但是这里我们可以用图画来更直观的展示数据的缺失情况。
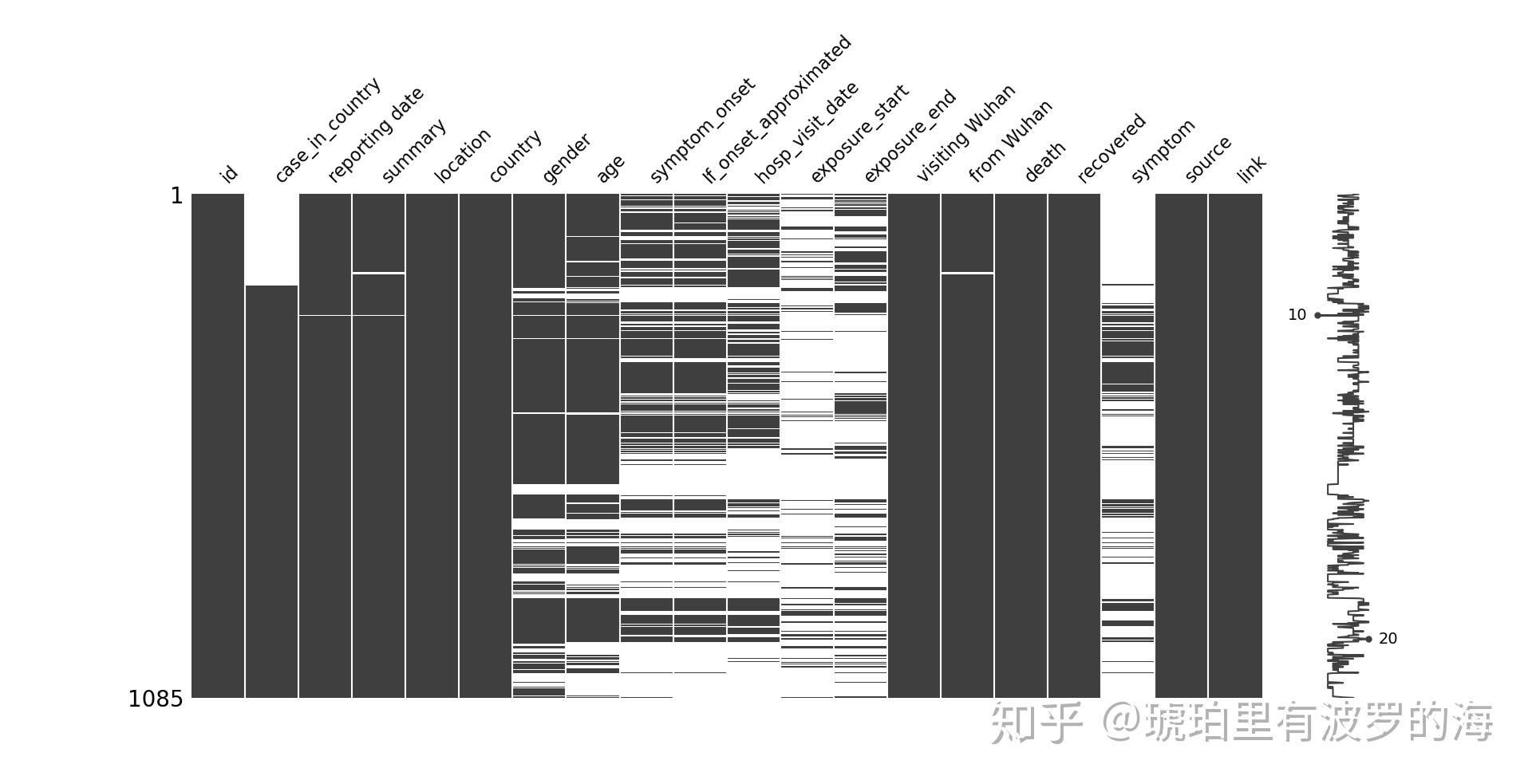
missingno 是专门进行缺失数据可视化的python 库,它自带多个可视化类型,比如matrix,bar chart,dendrogram等。对于小样本量,matrix会是不错的选择,更大的数据量可以选用dendrogram。 关于该库更多的详情,请参考github:https://github.com/ResidentMario/missingno。
msno.matrix(df=line_list_data_raw_df, fontsize=16)结果如下:左侧栏(Y轴)是样本量,我们最多的样本量为1085个。横坐标是特征名称,因为我们的特征比较少,所以可以清晰的展示。黑色表示该特征样本齐全,白色间隙表示该特征缺失部分样本。可以看到case_in_country 有样本缺失,而且集中在开始。画面的右侧有一条曲线(sparkline),用于展示每个样本特征个数。比如有个数字10,表示该行只有10个特征,数字20表示最多的一个样本有20个特征。
花式填充数据
数据清理的很关键的一种就是数据填充,下面我们就要针对不同的列进行填充,文中用的填充思路可能不是最佳的,但是目的是为了展示不同的填充方法的实现形式。我们不会简单的一根筋,只会填充为常数,均值或者其他统计指标。
时间格式的转换
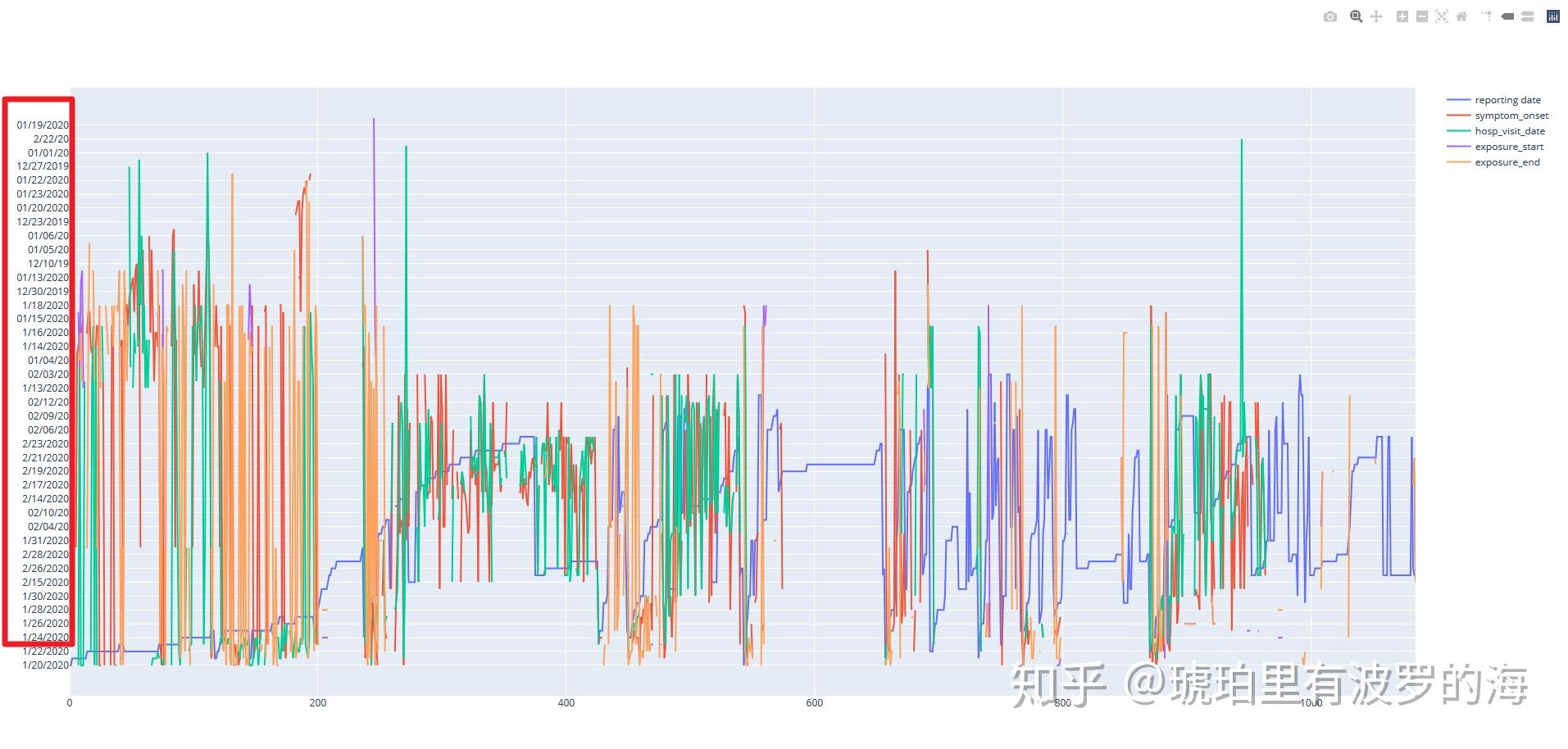
我们注意到有几列是时间相关的特征,我们首先要将其转成时间格式,python的时间格式很多,由于我们后续操作都用pandas,因此我这里将其转为pandas中的时间格式(Timestamp)。 我们可以先看一下不转时间格式,曲线图效果如何。我们采用plotly 画图,具体看代码。为什么用plotly? 因为可以交互!!
fig = go.Figure()
for col in date_cols:
fig.add_trace(go.Scatter(y=line_list_data_raw_df[col], name=col))
fig.show()可以看到Y坐标(红色框内所示)乱成一团。
我们查看一下这几列的数据格式有哪些。
date_cols = [
'reporting date',
'symptom_onset',
'hosp_visit_date',
'exposure_start',
'exposure_end']
print(line_list_data_raw_df[date_cols].head(5))
print(line_list_data_raw_df[date_cols].tail(5))可以看到结果中时间格式有多种,有的是1/20/2020, 有的是01/03/20,还有很多是NaN缺失。
reporting date symptom_onset hosp_visit_date exposure_start exposure_end
0 1/20/2020 01/03/20 01/11/20 12/29/2019 01/04/20
1 1/20/2020 1/15/2020 1/15/2020 NaN 01/12/20
2 1/21/2020 01/04/20 1/17/2020 NaN 01/03/20
3 1/21/2020 NaN 1/19/2020 NaN NaN
4 1/21/2020 NaN 1/14/2020 NaN NaN
reporting date symptom_onset hosp_visit_date exposure_start exposure_end
1080 2/25/2020 NaN NaN NaN NaN
1081 2/24/2020 NaN NaN NaN NaN
1082 2/26/2020 NaN NaN NaN 2/17/2020
1083 2/25/2020 NaN NaN 2/19/2020 2/21/2020
1084 2/25/2020 2/17/2020 NaN 2/15/2020 2/15/2020我们可以写一个小的函数来看一下时间数据的长度分布:
# check the length of date
for col in date_cols:
date_len = line_list_data_raw_df[col].astype(str).apply(len)
date_len_ct = Counter(date_len)
print(f'{col} datetiem length distributes as {date_len_ct}')可以看到时间字符串的长度不同,其中hosp_visit_date的长度有4种(除去长度为3的NaN)。
reporting date datetiem length distributes as Counter({9: 894, 8: 190, 3: 1})
symptom_onset datetiem length distributes as Counter({3: 522, 9: 379, 8: 167, 10: 17})
hosp_visit_date datetiem length distributes as Counter({3: 578, 9: 375, 8: 128, 10: 2, 7: 2})
exposure_start datetiem length distributes as Counter({3: 957, 9: 91, 8: 30, 10: 7})
exposure_end datetiem length distributes as Counter({3: 744, 9: 292, 8: 46, 10: 3})对于一般的字符串转成时间格式,pandas中to_datetime 函数可以解决问题,但是本案例中出现了mix的时间格式,因此我们需要一点小技巧来完成格式转换。
def mixed_dt_format_to_datetime(series, format_list):
temp_series_list = []
for format in format_list:
temp_series = pd.to_datetime(series, format=format, errors='coerce')
temp_series_list.append(temp_series)
out = pd.concat([temp_series.dropna(how='any')
for temp_series in temp_series_list])
return out代码核心思想:to_datetime 每次只能转一个时间格式,我们需要将格式不匹配的数据设置为NaT(没有笔误,不是NaN)。对于同一列,我们用不同的时间格式多次转换,最后求交集。或者你可以对每一行的数据进行分别判断,但是这个循环次数可能会比较多,我预测效率不是很高。
调用函数,转换时间格式,然后我们再次print info。可以看到数据的格式已经变成了datetime64[ns],表明转换成功。
for col in date_cols:
line_list_data_raw_df[col] = mixed_dt_format_to_datetime(
line_list_data_raw_df[col], ['%m/%d/%Y', '%m/%d/%y'])
print(line_list_data_raw_df[date_cols].info())
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 reporting date 1084 non-null datetime64[ns]
1 symptom_onset 563 non-null datetime64[ns]
2 hosp_visit_date 506 non-null datetime64[ns]
3 exposure_start 128 non-null datetime64[ns]
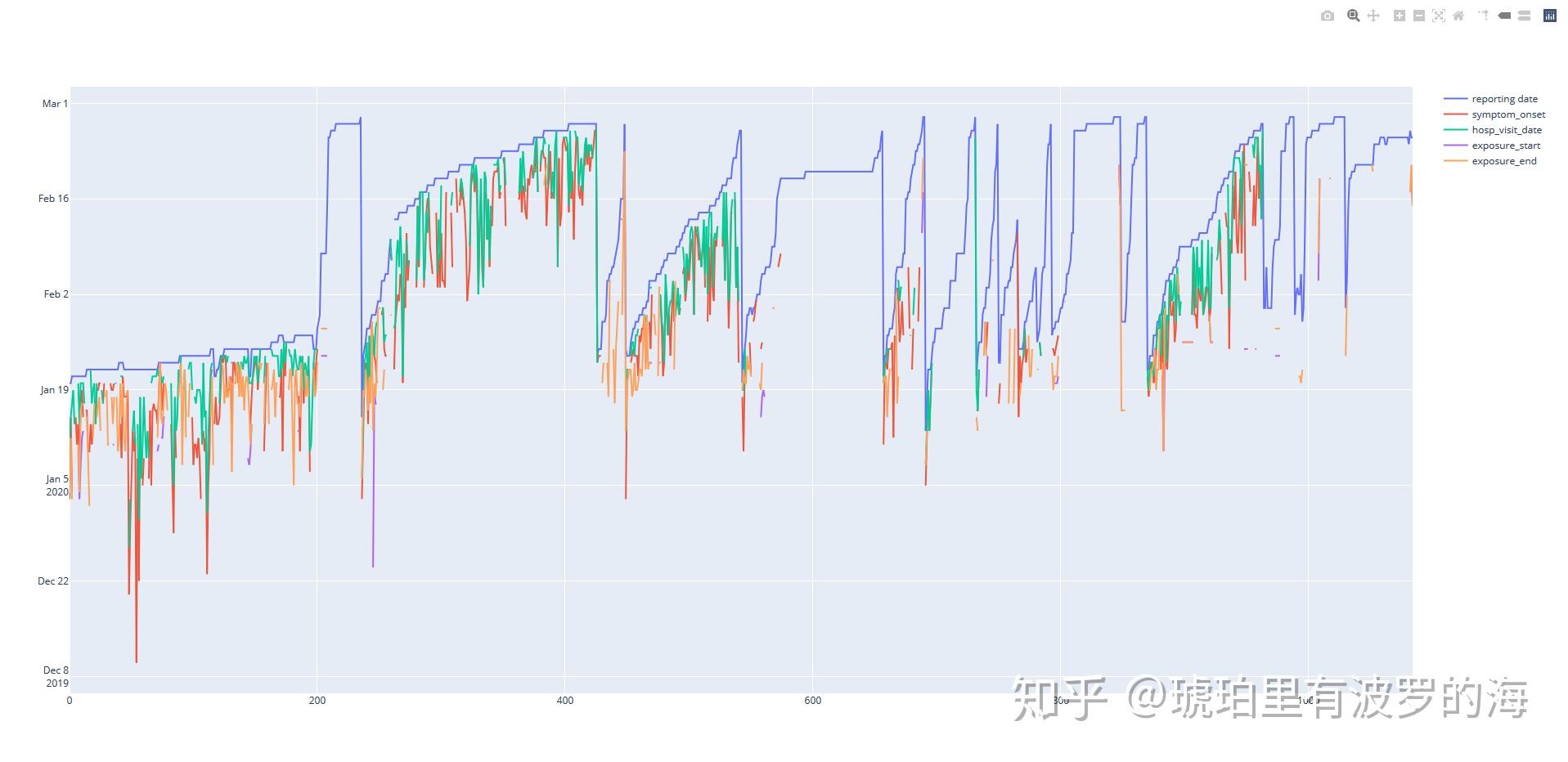
4 exposure_end 341 non-null datetime64[ns]此时我们可以再次plot 这几个曲线,Y轴已经变成很有条理的时间线。
- 我们观察该曲线,可以看到report_date曲线在最上端,也就是最晚的时间,这很符合逻辑。
- hospitalize_date 住院时间如果缺失的话,我们可以直接用报告时间代替。
- 根据逻辑,一般病人在有症状后,会隔一段时间或者立马去医院。因此hospitalize_date 必定会晚于symptom_onset 时间。这里我们可以做出统计看看病人有症状后多久会去医院,并以此为依据倒推symptom_onset时间。
- 与此类似,我们可以统计有暴露史的起始时间与病人发病的时间差,因此填充exposure_start。
- 至于exposure_end的缺失值,我们有理由相信,病人入院就结束暴露史。
以上就是我们的填充思路,具体的代码(技巧)如下:
直接赋值填充
# fill missing report_date
print(line_list_data_raw_df[pd.isnull(
line_list_data_raw_df['reporting date'])].index)
print(line_list_data_raw_df['reporting date'].iloc[260:263])
line_list_data_raw_df.loc[261, 'reporting date'] = pd.Timestamp('2020-02-11')
print(line_list_data_raw_df.info())根据其他列的信息填充
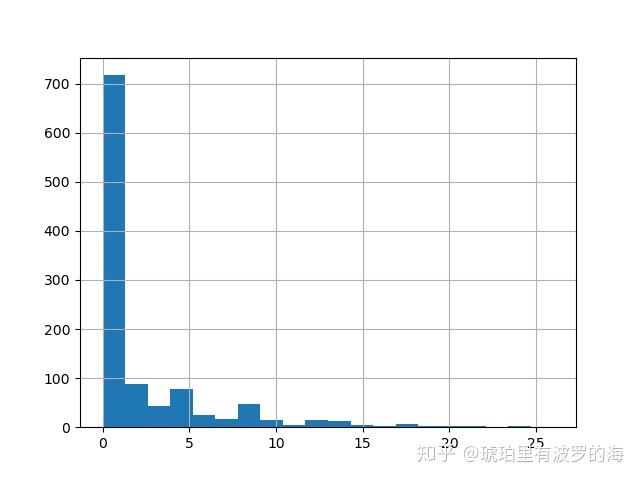
time_delta = line_list_data_raw_df['reporting date'] - \
line_list_data_raw_df['hosp_visit_date']
time_delta.dt.days.hist(bins=20)
line_list_data_raw_df['hosp_visit_date'].fillna(
line_list_data_raw_df['reporting date'], inplace=True)我们可以看到病人住院和报道的时间差(天数)分布,大部分还是在一天左右。所以我们可以近似的用reporting date的数据填充hosp_visit_date。
根据多列的信息推断填充
#fill missing symptom_onset
time_delta = line_list_data_raw_df['hosp_visit_date'] - \
line_list_data_raw_df['symptom_onset']
time_delta.dt.days.hist(bins=20)
average_time_delta = pd.Timedelta(days=round(time_delta.dt.days.mean()))
symptom_onset_calc = line_list_data_raw_df['hosp_visit_date'] - \
average_time_delta
line_list_data_raw_df['symptom_onset'].fillna(symptom_onset_calc, inplace=True)
print(line_list_data_raw_df.info())同样的,我们可以看看住院和病人有症状的时间差分布。这次分布最高点不再是1天附近,而是3天。也就是说大部分人在有症状之后3天左右的时间去医院,也有人接近25天才去。所以我们这里采用求均值的方法,然后根据入院时间倒推发病时间。
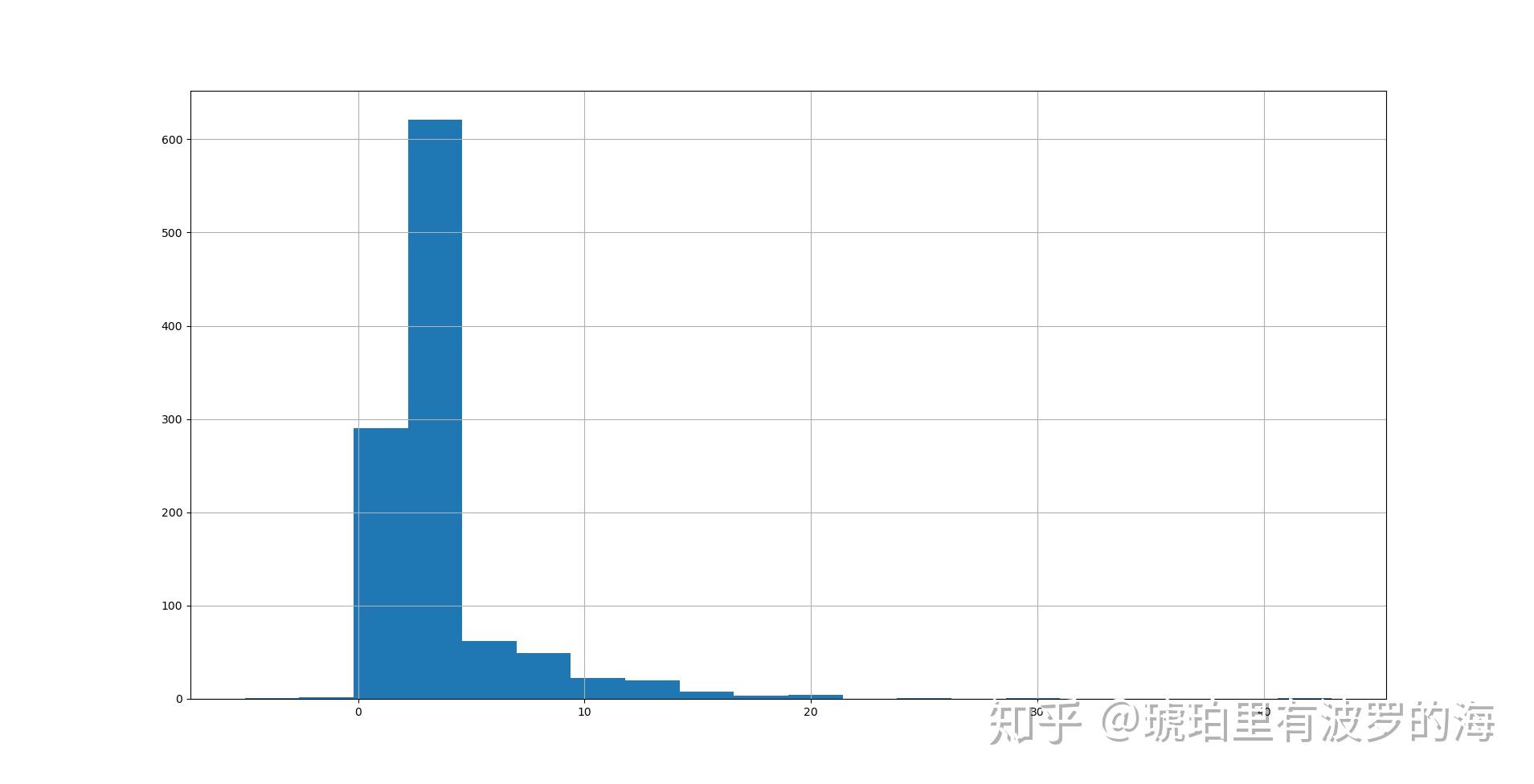
#fill missing exposure_start
time_delta = line_list_data_raw_df['symptom_onset'] - \
line_list_data_raw_df['exposure_start']
time_delta.dt.days.hist(bins=20)
average_time_delta = pd.Timedelta(days=round(time_delta.dt.days.mean()))
symptom_onset_calc = line_list_data_raw_df['symptom_onset'] - \
average_time_delta
line_list_data_raw_df['exposure_start'].fillna(symptom_onset_calc, inplace=True)
print(line_list_data_raw_df.info())大部分人有暴露史后,4天到10天内出现症状的概率较高,这也就是所谓的潜伏期。同理,我们可以以此倒推出暴露(感染)日期。
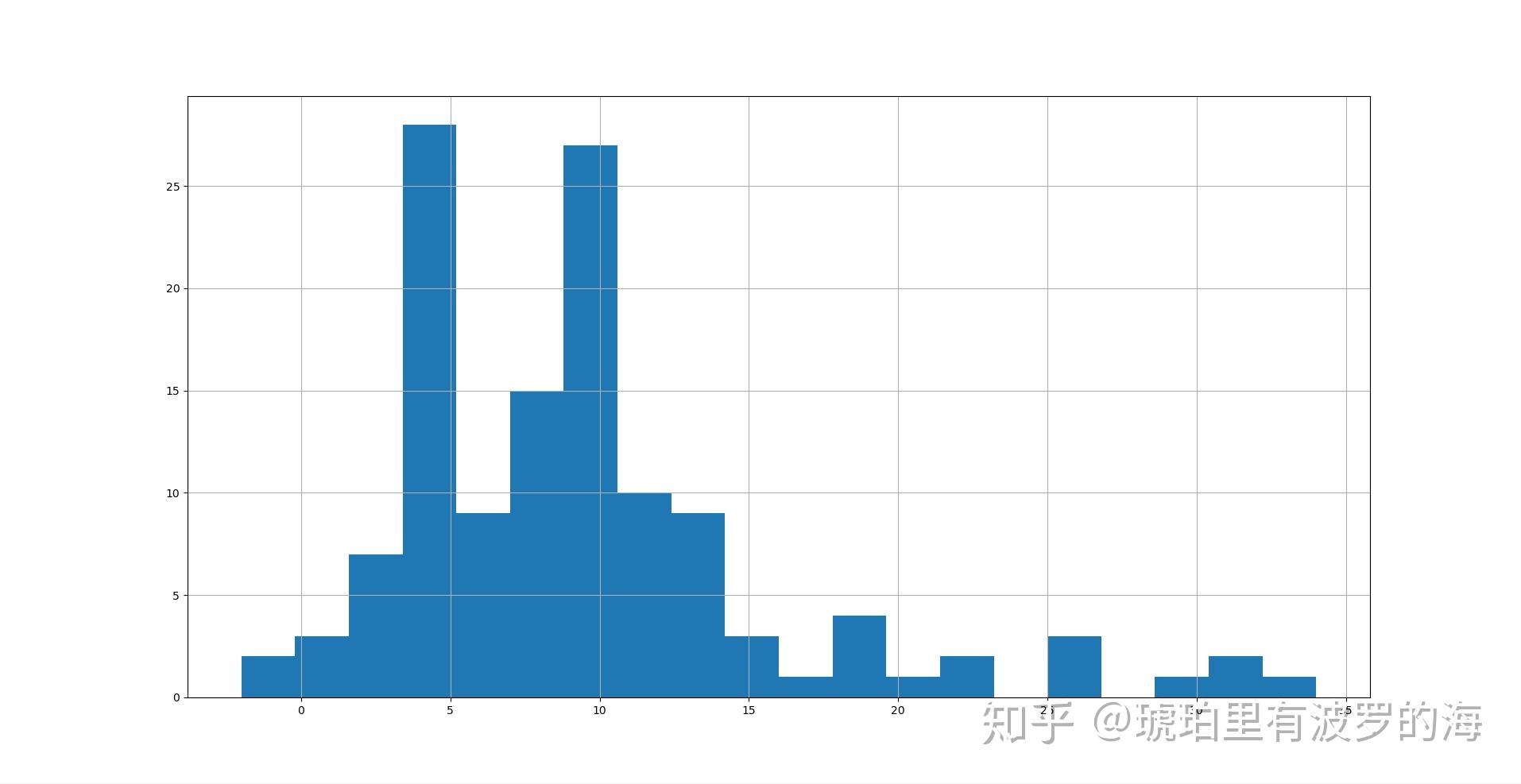
#fill missing exposure_end
line_list_data_raw_df['exposure_end'].fillna(line_list_data_raw_df['hosp_visit_date'], inplace=True)
print(line_list_data_raw_df.info())我们再次plot 这几个时间特征,可以看到他们已经没有缺失值。
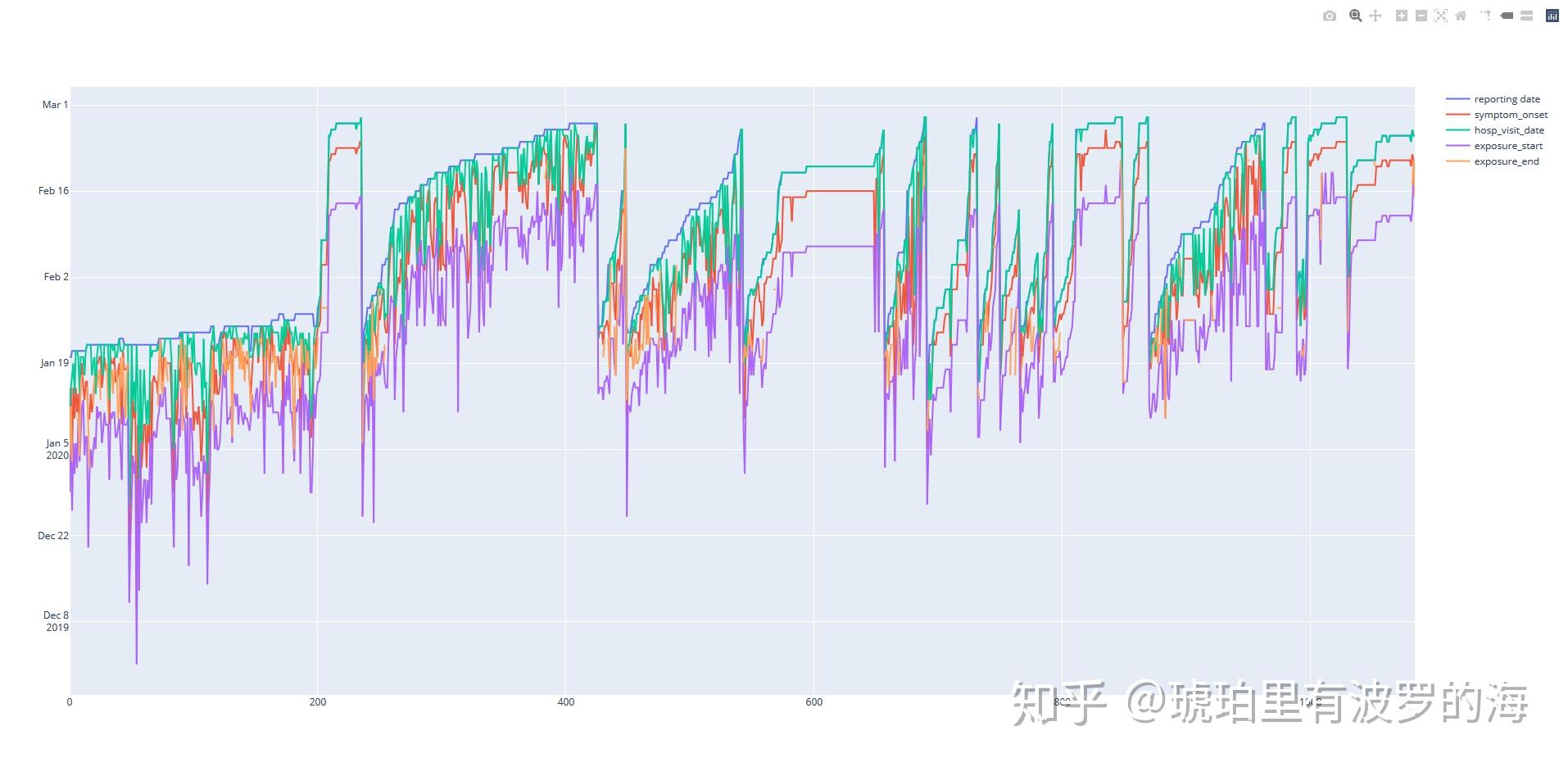
其他填充方法
其他的填充方法,思路见代码注释。
# case_in_country 在其他数据集中比较齐全,对于该数据集不重要,所以用-1 填充
line_list_data_raw_df['case_in_country'].fillna(-1, inplace=True)
print(line_list_data_raw_df.info())
# summary 每个case 都不相同,无法推断,因此替换为空字符串
print(line_list_data_raw_df['summary'].head(5))
line_list_data_raw_df['summary'].fillna('', inplace=True)
# 虽然性别可以统计,但是这里我们直接用unknown 代替
print(line_list_data_raw_df.info())
print(line_list_data_raw_df['gender'].head(5))
line_list_data_raw_df['gender'].fillna('unknown', inplace=True)
# 年龄采用均值代替
line_list_data_raw_df['age'].hist(bins=10)
line_list_data_raw_df['age'].fillna(
line_list_data_raw_df['age'].mean(), inplace=True)
line_list_data_raw_df['age'].hist(bins=10)
# If_onset_approximated 设为1表示都是我们猜测的
print(line_list_data_raw_df['If_onset_approximated'].head(5))
line_list_data_raw_df['If_onset_approximated'].fillna(1, inplace=True)
print(line_list_data_raw_df.info())
# from Wuhan 丢失的数据在index 166和175 之间,可以看到location 是北京,而且属于早期,因此我们可以设为1,表示来自武汉。
print(line_list_data_raw_df[pd.isnull(
line_list_data_raw_df['from Wuhan'])].index)
print(line_list_data_raw_df[['from Wuhan','country','location']].iloc[166:175])
line_list_data_raw_df['from Wuhan'].fillna(1.0,inplace=True)
# 我们通过统计词频,选取出现最高的symptom 来代替缺失值。可以看到最常见的symtom 是发烧。
symptom = Counter(line_list_data_raw_df['symptom'])
print(symptom.most_common(2)[1][0])
line_list_data_raw_df['symptom'].fillna(symptom.most_common(2)[1][0],inplace=True)再次查看缺失matrix,bingo!虽然matrix不再花哨(黑白相间),但是这是最完美的黑。
# missing data visualization
msno.matrix(df=line_list_data_raw_df, fontsize=16)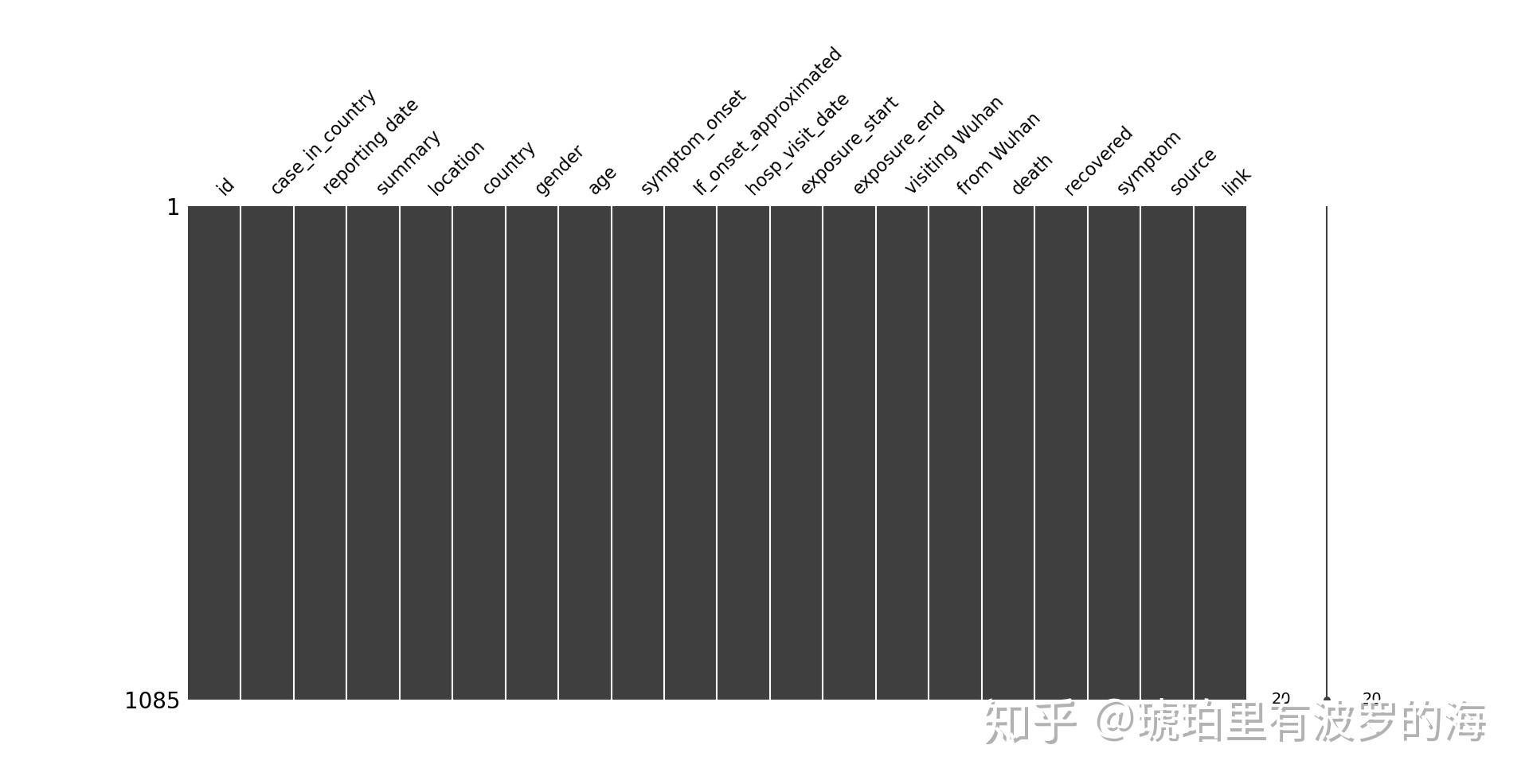
总结
本文中主要介绍了数据清理尤其是填充相关的技巧。你可以填充一个具体的值,空值,统计值或者是根据其他的列进行推断。
其中也涉及到一些小技巧,比如混合的时间格式如何转成datetime,如何对数据缺失情况进行可视化。
我们没有对该数据进行EDA处理,但是在数据清理的过程中,我们还是对该病程有了一点更多的了解: 比如病人潜伏期在4天到10天比较多,病人出现症状后一般3天左右去医院,症状最多的是发烧,等等。

